Skillnad mellan misshandelsreparation och nukleotidreparationsreparation | Mismatch Repair vs Nucleotide Excision Repair
Huvudskillnad - Mismatch Repair vs Nucleotide Excision Repair
Tent och tusentals DNA-skador uppstår i cellen per dag. Det inducerar förändringar i cellprocesserna såsom replikation, transkription såväl som cellens livskraft. I vissa fall kan mutationer som orsakas av dessa DNA-skador leda till skadliga sjukdomar som cancer och åldringsrelaterade syndrom (ex: Progeria). Oavsett dessa skador initierar cellen en högorganiserad kaskadreparationsmekanism som kallas DNA-skador. Flera DNA-reparationssystem har identifierats i cellsystemet; Dessa är kända som Base Excision Repair (BER), Mismatch Repair (MMR), Nucleotide Excision Repair (NER), Double Strand Break Repair. Nukleotidutjämningsreparation är ett mycket mångsidigt system som känner igen skrämmande helixförvrängning av DNA-lesioner och tar bort dem. Å andra sidan ersätter mismatch reparation felaktiga baser under replikering. Den viktigaste skillnaden mellan mismatchreparation och nukleotidutjämningsreparation är att nukleotidutjämningsreparation (NER) används för att avlägsna pyrimidindimerer som bildas genom UV-bestrålning och skrymmande helix-lesioner orsakade av kemiska addukter medan felparametrarna spelar en viktig roll för att korrigera felaktigt baser som har rymt från replikationsenzymer (DNA-polymeras 1) under postreplikering. Förutom felaktiga baser kan MMR-systemproteiner också reparera insats / deletionsslingorna (IDL) som är resultat av polymerasglidning under replikation av repetitiva DNA-sekvenser.
INNEHÅLL
1. Översikt och nyckelfaktor
2. Vad är Mismatch Repair
3. Vad är Nucleotide Excision Repair
4. Jämförelse vid sida vid sida - Mismatch Repair vs Nucleotide Excision Repair
5. Sammanfattning
Vad är Nucleotide Excision Repair?
Den mest framträdande egenskapen vid nukleotidutjämningsreparation är att den reparerar de modifierade nukleotidskadorna som orsakas av signifikanta distorsioner i DNA-dubbelhelixen. Det observeras i nästan alla organismer som har undersökts aktuellt. Uvr A, Uvr B, Uvr C (excinucleases) Uvr D (ett helikas) är de mest kända enzymerna som är involverade i NER, vilket utlöser reparationen av DNA i modellorganismen Ecoli. Uvr ABC multi-subenheter enzymkomplex producerar Uvr A, Uvr B, Uvr C polypeptider.Generna kodade för ovan nämnda polypeptider är uvr A, uvr B, uvr C. Uvr A och B enzymer känner kollektivt igen av den skadainducerad förvrängning som orsakas av DNA-dubbel-helixen, såsom pyrimidindimmare på grund av UV-bestrålning. Uvr A är ett ATPase-enzym och detta är en autokatalytisk reaktion. Därefter lämnar Uvr A DNA medan Uvr BC-komplex (aktiv nukleas) klyver DNA i båda sidor av skadan som katalyseras av ATP. Ett annat protein som heter Uvr D som kodas av uvrD-genen är ett helikas II-enzym som släpper av det DNA som härrör från frigörandet av enkelsträngat skadat DNA-segment. Detta lämnar ett gap i DNA-helixen. Efter skadad segment har skurits, kvarstår ett 12-13 nukleotidspalt i DNA-strängen. Detta fylls upp av DNA-polymerasenzymet I och nicket förseglas av DNA-ligasen. ATP krävs vid tre steg av denna reaktion. NER-mekanismen kan också identifieras hos däggdjursliknande människor. Hos människor är hudtillståndet Xeroderma pigmentosum beror på DNA-dimerer som orsakas av UV-bestrålning. Generna XPA, XPB, XPC, XPD, XPE, XPF och XPG producerar proteiner som ersätter DNA-skador. Proteinerna från gener XPA, XPC, XPE, XPF och XPG har nukleasaktiviteten. Å andra sidan visar proteinerna i XPB och XPD-gener helikasaktiviteten som analoger med Uvr D i E coli.

Figur 01: Nucleotid Excisionsreparation
Vad är Mismatch Repair?
Felparametrar reparationssystemet initieras under DNA-syntesen. Även med funktionell € -underenheten möjliggör DNA-polymeras III inkorporeringen av en fel nukleotid för syntesen varje 10 8 baspar. Mismatch reparationsproteiner känner igen denna nukleotid, excise den och ersätta den med den korrekta nukleotiden som är ansvarig för den slutliga graden av noggrannhet. DNA-metylering är pivotal för MMR-proteiner för att igenkänna föräldersträngen från den nyligen syntetiserade strängen. Metyleringen av adenin (A) nukleotid i ett GATC-motiv av en nysyntetiserad sträng är lite fördröjd. Å andra sidan har modersträngadadenukleotiden i GATC-motiv redan metylerat. MMR-proteiner känner igen den nyssyntetiserade strängen med denna skillnad från modersträngen och startar mismatch reparation i en nysyntetiserad sträng innan den blir metylerad. MMR-proteinerna dirigerar sin reparationsaktivitet för att excisera fel nukleotid innan den nyligen replikerade DNA-strängen blir metylerad. Enzymerna Mut H, Mut L och Mut S kodade av gener mut H, mut L, mut S katalyserar dessa reaktioner i Ecoli. Mut S-protein känner igen sju av åtta möjliga mismatchbaspar, förutom för C: C, och binder vid platsen för otillbörlig matchning i duplex-DNA. Med bundna ATP sätter Mut L och Mut S sig i komplexet senare. Komplexet translokerar några tusen baspar bort tills det finner en hemimetylerad GATC-motiv. Den sovande nukleasaktiviteten hos Mut H-protein aktiveras när den finner en hemimetylerad GATC-motiv. Det klyver den icke-metylerade DNA-strängen som lämnar ett 5'-nick vid G-nukleotiden av osmetylerat GATC-motiv (nysyntetiserad DNA-sträng).Därefter nickas samma sträng på den andra sidan av mismatchen av Mut H. I de övriga stegen avkänner kollektiva verkningarna av Uvr D ett helikasprotein, Mut U, SSB och exonukleas I den felaktiga nukleotiden i den enkelsträngade DNA. Spalten som bildas i excisionen fylls upp av DNA-polymeras III och förseglas genom ligas. Ett liknande system kan identifieras hos möss och människor. Mutationen av humant hMLH1, hMSH1 och hMSH2 är involverade i ärftlig icke-polypos tjocktarmscancer som avreglerar celldelning av kolonceller.
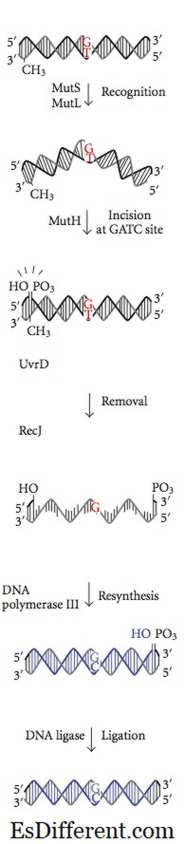
Figur 02: Mismatch Repair
Vad är skillnaden mellan Mismatch Repair och Nucleotide Excision Repair?
- diff Artikel Middle before Table ->
Mismatch Reparation vs Nucleotide Excision Repair |
|
| Avvikande reparationssystem inträffar under efter replikering. | Detta är inblandat i avlägsnande av pyrimidindimerer på grund av UV-bestrålning och andra DNA-lesioner på grund av kemisk addukt. |
| Enzymer | |
| Det katalyseras av Mut S, Mut L, Mut H, Uvr D, SSB och exonukleas I. | Det katalyseras av Uvr A, Uvr B, Uvr C, UvrD enzymer. |
| Metylering | |
| Det är pivotalt att initiera reaktionen. | DNA-metylering krävs inte för att initiera reaktionen. |
| Enzymernas verkan | |
| Mut H är ett endonukleas. | Uvr B och Uvr C är exonukleaser. |
| Event | |
| Detta händer specifikt under replikering. | Detta sker vid exponering för U.V eller kemiska mutagener, inte under replikering |
| Bevarande | |
| Den är mycket konserverad | Den är inte starkt konserverad. |
| Gap-fyllning | |
| Det görs genom DNA-polymeras III. | Det görs av DNA-polymeras I. |
Sammanfattning - Mismatch Repair vs Nucleotide Excision Repair
Mismatch reparation (MMR) och Nucleotide Excision Repair (NER) är två mekanismer som äger rum i cellen för att rätta till DNA-skador och förvrängningar som orsakas av olika agenter. Dessa kallas kollektivt som DNA-reparationsmekanismer. Nukleotidutjämningsreparation reparerar de modifierade nukleotidskadorna, typiskt de signifikanta skadorna av DNA-dubbelhelixen som händer på grund av exponering för UV-bestrålning och kemiska addukter. Mismatch reparationsproteiner känner igen fel nukleotid, excise det och ersätta den med korrekt nukleotid. Denna process är ansvarig för den slutliga graden av noggrannhet under replikering.
Referens:
1. Cooper, Geoffrey M. "DNA Reparation. "Cellen: En molekylär tillvägagångssätt. 2: a upplagan. U. S. National Library of Medicine, 01 jan 1970. Web. 09 mars 2017.
2. "Mekanismer och funktioner för DNA-mismatch reparation. "Cellforskning. U. S. National Library of Medicine, n. d. Webb. 09 mars 2017.
Image Courtesy:
1. "Nucleotide Excision Repair-journal. pbio. 0040203. g001 "Av Jill O. Fuss, Priscilla K. Cooper - (CC BY 2. 5) via Commons Wikimedia
2. "DNA mismatch repair Ecoli" Av Kenji Fukui - (CC BY 4. 0) via Commons Wikimedia




